ABSTRACT
Objective: The aim of this study was to determine the prevalence of macrolide-resistant S. pneumoniae and to identify its phenotypic and genotypic characteristics. Methods: Strains of S. pneumoniae isolated in the city of Porto Alegre between May 2002 and August 2004 from samples collected from different anatomical sites were analyzed. For the agar diffusion test, disks of erythromycin, clarithromycin, azithromycin and clindamycin were used. The minimum inhibitory concentrations of erythromycin were determined for macrolide-resistant isolates by the agar dilution method. Macrolide-resistant isolates were phenotyped by agar diffusion test and genotyped by polymerase chain reaction. Results: A total of 229 pneumococcal strains were evaluated, 12 (5.2%) of which were macrolide-resistant. Among the 12 resistant strains, 9 (75%) presented the MLSB phenotype, and 3 (25%) presented the M phenotype. Polymerase chain reaction testing indicated that 8 MLSB phenotype isolates harbored the ermB gene only, whereas the mefE gene was present in all 3 M phenotype isolates. One MLSB phenotype isolate presented both genes. Conclusion: In Porto Alegre, the S. pneumoniae resistance to macrolides is still low since such resistance is due primarily to the presence of the ermB gene expressing the MLSB phenotype.
RESUMO
Objetivo: O objetivo deste estudo foi determinar a prevalência do S. pneumoniae resistente aos macrolídeos e identificar suas características fenotípicas e genotípicas. Métodos: Amostras de S. pneumoniae isoladas entre maio de 2002 e agosto de 2004, em Porto Alegre (RS), a partir de materiais clínicos coletados de diferentes sítios anatômicos foram analisadas. Para o teste de difusão em ágar foram utilizados discos de eritromicina, claritromicina, azitromicina e clindamicina. As concentrações inibitórias mínimas de eritromicina foram determinadas nos isolados resistentes aos macrolídeos pelo método de diluição em ágar. Os fenótipos dos isolados resistentes aos macrolídeos foram investigados pelo teste de difusão em ágar e a genotipagem pela reação em cadeia da polimerase. Resultados: Foram avaliados 229 isolados de pneumococos, e 12 mostraram-se resistentes aos macrolídeos (5,2%). Entre estes, 9 apresentaram o fenótipo MLSB (75%) e 3 o fenótipo M (25%). A reação em cadeia da polimerase indicou que 8 isolados com o fenótipo MLSB portavam apenas o gene ermB, enquanto que o gene mefE estava presente em todos os 3 isolados com o fenótipo M. Um isolado com o fenótipo MLSB apresentou ambos os genes. Conclusão: A resistência aos macrolídeos do S. pneumoniae em Porto Alegre permanece baixa, sendo devida principalmente à presença do gene ermB, com expressão do fenótipo MLSB.
INTRODUÇÃOCom o crescimento da resistência do Streptococcus pneumoniae à penicilina nas últimas décadas, tem sido necessário o uso de drogas alternativas como os macrolídeos.(1) Entretanto, recentemente também tem sido observado um aumento significativo da resistência do pneumococo a esta classe antimicrobiana em vários países do mundo.(2-4)
A resistência do S. pneumoniae aos macrolídeos ocorre devido a dois mecanismos principais: modificação do sítio de ligação da droga e efluxo ativo. O primeiro ocorre devido à aquisição do gene ermB, o qual confere resistência aos macrolídeos, lincosamídeos e estreptogramina B, sendo esta resistência múltipla, o que dá o nome ao fenótipo: MLSB. O mecanismo de resistência ligado ao efluxo ativo está associado ao gene mefE, que confere resistência somente aos membros catorze e quinze dos macrolídeos (eritromicina, claritromicina e azitromicina), resultando no fenótipo M.(5-7)
Variações na prevalência desses genes e mecanismos de resistência aos macrolídeos têm sido verificados entre pneumococos isolados em diferentes regiões do mundo.(8-9) O objetivo deste estudo é investigar a ocorrência, os mecanismos mo-leculares e a expressão fenotípica da resistência aos macrolídeos em amostras de pneumococos oriundas de diversos hospitais de Porto Alegre (RS).
MÉTODOSForam analisadas inicialmente 259 amostras de S. pneumoniae isoladas a partir de diversos materiais clínicos. Essas amostras foram obtidas no período de maio de 2002 a agosto de 2004, nos hospitais colaboradores do estudo: Hospital Mãe de Deus, Hospital São Lucas da Pontifícia Universidade Católica do Rio Grande do Sul, Hospital Nossa Senhora da Conceição, Irmandade Santa Casa de Misericórdia de Porto Alegre e Hospital de Clínicas de Porto Alegre.
A identificação das amostras foi confirmada utilizando-se a determinação do tipo de hemólise, a morfologia colonial e a suscetibilidade à optoquina.(10)
Todos os isolados clínicos foram testados pelo método de difusão em ágar (TDA), utilizando-se discos de eritromicina de 5 µg (Oxoid Ltd, England), claritromicina de 2 µg (Oxoid Ltd, England), azitromicina de 15 µg (Oxoid Ltd, England) e clindamicina de 2 µg (Oxoid Ltd, England). A suspensão bacteriana foi semeada, com o auxílio de um swab estéril, sobre a superfície de uma placa de ágar Mueller-Hinton suplementada com 5% de sangue de carneiro (MHS; bioMérieux, Marcy l´Etoile, France). Os discos de eritromicina, claritromicina, azitromicina e clindamicina foram colocados sobre a superfície do meio de cultura com o auxílio de uma pinça esterilizada. As placas foram incubadas a 35oC por 20 a 24h em atmosfera de CO2. Quando não foram constatados halos de inibição ao redor dos discos de macrolídeos e clindamicina, o fenótipo foi considerado MLSB. Quando não foram constatados halos de inibição ao redor dos discos de macrolídeos e halo de sensibilidade ao redor do disco de clindamicina, o fenótipo foi considerado M.
As amostras resistentes aos macrolídeos pelo TDA (teste de triagem) foram testadas para determinação de suas concentrações inibitórias mínimas (CIM) através do teste de diluição em ágar para eritromicina. A 17 ml do meio Mueller-Hinton foram acrescidos 2 ml de cada diluição do antimicrobiano e 1 ml de sangue desfibrinado de carneiro. Foram preparados meios contendo concentrações conhecidas de eritromicina (0,25 µg/ml a 8 µg/ml). As placas contendo as diferentes concentrações do antimicrobiano foram semeadas com auxílio de um replicador do tipo Steers, utilizando-se uma suspensão de S. pneumoniae diluída a 1:10 em solução salina estéril. O inóculo final, depositado sobre a superfície das placas, foi de aproximadamente 104 unidades formadoras de colônias/ml. As placas foram incubadas a 35oC por 20 a 24h, em atmosfera com 5% de CO2. Os resultados das CIM obtidas foram interpretados conforme o National Committee for Clinical Laboratory Standards, de 2004.(11)
As amostras resistentes fenotipicamente foram testadas através da reação em cadeia da polimerase para a presença dos genes ermB e mefE, conforme protocolo introduzido por Sutcliffe et al.(12) Para confirmar a especificidade da reação em cadeia da polimerase, foram também testadas amostras fenotipicamente suscetíveis de pneumococo, escolhidas aleatoriamente entre todas as que foram sensíveis aos macrolídeos pelo TDA.
A extração do DNA foi realizada através da ressuspensão de colônias isoladas de S. pneumoniae retiradas do meio de cultura em 300 µl de tampão fosfato salina, seguida de centrifugação a 3.000 rpm por quinze minutos. O sobrenadante foi desprezado e o sedimento foi utilizado para a extração do DNA. O sedimento foi ressuspenso em 50 µl de tampão Tris-EDTA 1x (TE) pH 7,4, incubado por dez minutos a 37oC e a 100oC por três minutos. As amostras foram mantidas congeladas a -20°C até a sua utilização (entre um e três dias).
A reação em cadeia da polimerase foi realizada em um volume total de 50 l contendo 200 µM de desoxinucleotídeos trifosfatados (dATP, dCTP, dTTP, dGTP), 1,4 µM do iniciador (ermB direto: 5' - GAA AAG GTA CTC AAC CAA - 3'; ermB reverso: 5' - AGT AAC GGT ACT TAA ATT GTT - 3'; mefE direto: 5' - AGT ATC ATT AAT CAC TAG TGC - 3'; mefE reverso: 5 '-TTC TTC TGG TAC TAA AAG TGG - 3'), 0,2 U de Taq DNA polimerase, 10 mM de Tris-HCl (pH 8,3), e 1 µl do DNA extraído. Para detecção do gene ermB foi utilizada a concentração de 2 mM de cloreto de magnésio e para detecção do gene mefE 4 mM.
RESULTADOSDas 259 amostras inicialmente encaminhadas pelos hospitais participantes do estudo, 30 foram excluídas por morte bacteriana. As 229 amostras de S. pneumoniae restantes eram provenientes de diversos materiais clínicos, a saber: sangue, 80 amostras; escarro, 75 amostras; líquor, 20 amostras; líquido pleural, 15 amostras; aspirado traqueal, 12 amostras; secreção ocular, 11 amostras; secreção brônquica, 7 amostras; outros materiais, 9 amostras.
Todos os isolados clínicos foram testados pelo TDA, utilizando-se discos impregnados com antimicrobianos (eritromicina, claritromicina e azitromicina). Os resultados da suscetibilidade aos macrolídeos estão apresentados na Tabela 1.
Os isolados clínicos que mostraram resistência pelo TDA foram testados para a CIM de eritromicina pelo teste de diluição em ágar. Os resultados estão apresentados na Tabela 2.
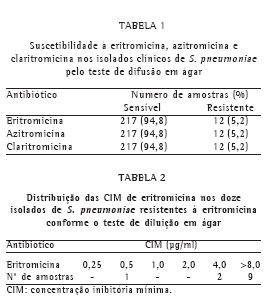
Dos 12 isolados que foram testados pelo TDA para os macrolídeos e clindamicina e demonstraram resistência à eritromicina, 9 apresentaram o fenótipo MLSB (75%) e 3 o fenótipo M (25%).
Foram encontradas 8 amostras positivas para o gene ermB, 3 positivas para o gene mefE e 1 positiva para ambos. A Figura 1 representa a visualização do resultado da amplificação de cinco isolados de S. pneumoniae.

Foi encontrada uma relação entre os fenótipos e genótipos de resistência, ou seja, as amostras portadoras do gene mefE apresentaram, na maioria dos casos, o fenótipo M, e as amostras portadoras do gene ermB apresentaram o fenótipo MLSB.
DISCUSSÃOO surgimento e a disseminação de cepas de S. pneumoniae resistentes à penicilina e aos macrolídeos têm causado crescente preocupação em todo o mundo. Variações geográficas consideráveis nessa resistência têm sido observadas, genotípica e fenotipicamente, sendo fundamental a monitorização dos seus padrões locais, a fim de se orientar de maneira mais específica a terapêutica antimicrobiana adequada a cada região.
Em nosso estudo, a resistência fenotípica do S. pneumoniae aos macrolídeos, determinada pelo TDA, foi de 5,2%, para eritromicina, azitromicina e claritromicina. Esta prevalência de resistência é semelhante à verificada no único estudo realizado em Porto Alegre encontrado na literatura, em que foram analisadas 417 amostras de pneumococos, isoladas no período de 1995 a 1998, o qual encontrou 4,5% de resistência à eritromicina.(13) Resultados similares foram encontrados em outro trabalho, com amostras coletadas de 1990 a 1999 em Porto Alegre, São Paulo (SP) e Rio de Janeiro (RJ). Nos 931 isolados de S. pneumoniae observou-se uma prevalência de resistência de 4,3% à eritromicina.(14) No estudo PROTEKT, realizado de 1999 a 2000, que incluiu países da América Latina (Argentina, Brasil e México), a resistência à eritromicina encontrada foi de 15,3%, sendo de 6,5% nas amostras brasileiras.(2) Um estudo mais recente, englobando os anos de 2001 e 2002, demonstrou taxas de resistência de S. pneumoniae de 9,5% para azitromicina e claritromicina em amostras coletadas em vários Estados brasileiros.(15)
Estudos realizados em outras regiões do mundo relatam dados de resistência bem mais elevados que no Brasil. No Asian Network for Surveillance of Resistant Pathogens, que envolveu dez países da Ásia com 555 isolados, realizado no período de 1998 a 2001, a resistência à eritromicina foi de 59,3%.(8) A resistência aos macrolídeos foi também avaliada na Alemanha, no período de 2002 a 2003, onde 241 amostras de pneumococos foram analisados e observaram-se 19,9% de cepas resistentes.(9) Uma prevalência de resistência semelhante, de 27,9%, ocorreu em outro levantamento do estudo PROTEKT, do qual participaram 46 países americanos, com um total de 10.102 amostras.(4)
Em nosso estudo, analisando-se as 12 amostras que foram resistentes aos macrolídeos pelo TDA, verificou-se que 9 delas apresentaram o fenótipo MLSB (75%) e 3 apresentaram o fenótipo M (25%). Em um trabalho realizado com 931 amostras de pneumococo isoladas em várias cidades brasileiras, foram encontradas 40 amostras resistentes à eritromicina (4,3%), sendo que 37 destas apresentaram o fenótipo MLSB (92,5%) e 3 o fenótipo M (7,5%).(14) Quando analisadas apenas as 13 amostras isoladas em Porto Alegre neste estudo, verificou-se que 2 apresentaram o fenótipo M (15,3%). Sendo assim, observou-se que a prevalência do fenótipo M em Porto Alegre foi semelhante à encontrada em nosso estudo.
Quando o valor da CIM de eritromicina foi avaliado nas amostras que foram resistentes aos macrolídeos, foram encontradas 9 amostras com CIM 8 g/ml, 2 com CIM de 4 g/ml e 1 amostra com CIM de 0,5 g/ml. Todas as amostras com CIM > 8 g/ml apresentaram o fenótipo MLSB e as amostras com CIM de 4 g/ml e 0,5 g/ml, o fenótipo M. As CIM de eritromicina também se relacionaram com a presença dos genes testados, ou seja, as amostras portadoras do gene ermB apresentaram CIM 8 g/ml e as portadoras do gene mefE tiveram CIM mais baixas, variando entre 0,5 g/ml e 4 g/ml. Essa associação foi também verificada em um estudo com 124 amostras resistentes à eritromicina provenientes de Christchurch, Nova Zelândia, onde 117 delas (94,3%) apresentaram alto nível de resistência à eritromicina (CIM 128 g/ml) e 7 apresentaram CIM entre 4 g/ml e 8 g/ml. Quando se verificou a presença dos genes nessas mesmas amostras e associou-se essa presença com as CIM de eritromicina, 41 amostras portadoras do gene ermB apresentaram CIM 128 g/ml, e 6 portadoras do gene mefE tiveram CIM entre 4 g/ml e 8 g/ml, e, dos 77 isolados portadores de ambos os genes, 76 apresentaram CIM 128 g/ml.(16) O isolado do nosso estudo que foi portador de ambos os genes também mostrou alto nível de resistência à eritromicina.
Um trabalho realizado na Turquia, que estudou 326 amostras de pneumococo, encontrou 13,8% de amostras resistentes à eritromicina, sendo 87,5% delas portadoras do gene ermB e 12,5% do gene mefE.(17) Nos EUA, em um levantamento do estudo PROTEKT realizado entre 2001 e 2002, analisando 2.793 amostras de pneumococos resistentes à eritromicina, observou-se que 68,7% dos isolados possuíam o gene mefE, 16,8% o gene ermB e 12,2% ambos os genes.(4) No Brasil, em um estudo realizado com amostras do período de 1990 a 1999, encontraram-se 40 isolados de pneumococo resistentes à eritromicina, sendo que 92,5% deles possuíam o gene ermB e 7,5% o gene mefE.(14)
Com relação à análise genotípica realizada nos 12 isolados que foram resistentes fenotipicamente aos macrolídeos, nossos resultados mostraram que 8 amostras foram portadoras do gene ermB (66,6%), 3 do gene mefE (25%) e 1 amostra foi portadora de ambos os genes (8,33%).
Isolados portadores de ambos os genes (ermB e mefE) têm sido relatados em alguns países do mundo, como no estudo realizado nos EUA entre 1996 e 1997, que encontrou uma prevalência de 7%.(18) Alguns autores descreveram o sorotipo 19F, clone multirresistente com ambos os genes, em 30,5% dos isolados provenientes de cinco laboratórios da África do Sul.(19) Dos 1.043 isolados resistentes aos macrolídeos do estudo PROTEKT, realizado entre 1999 e 2000, provenientes na sua maioria da Coréia do Sul, 71 foram positivos para os genes ermB e mefE (6,8%).(20) No Brasil, o presente estudo foi o primeiro a identificar um isolado de S. pneumoniae portador de ambos os genes ermB e mefE.
Em nosso estudo, verificamos que houve uma relação entre a resistência fenotípica e a genotípica, ou seja, os 8 isolados que apresentaram o fenótipo MLSB eram portadores do gene ermB e os 3 isolados que apresentaram o fenótipo M eram portadores do gene mefE. O isolado que foi positivo para ambos os genes apresentou o fenótipo MLSB devido à resistência à clindamicina conferida pelo gene ermB. Isso também foi verificado em outro estudo, em que as 37 amostras portadoras do gene ermB apresentaram o fenótipo MLSB e as 3 amostras portadoras do gene mefE apresentaram o fenótipo M.(14)
O estudo da resistência genotípica é de extrema relevância, uma vez que nos isolados portadores do gene mefE os macrolídeos poderão ser utilizados na clínica mesmo nos casos de amostras resistentes in vitro, enquanto que nas amostras portadoras do gene ermB, devido ao alto nível de resistência in vitro, existe a possibilidade de falha terapêutica.
Dessa forma, é fundamental a contínua monitorização dos padrões locais de resistência através de estudos de vigilância epidemiológica, a fim de se evitar o uso indiscriminado de antimicrobianos e o conseqüente crescimento e disseminação da resistência.
REFERÊNCIAS 1. Bartlett JG, Dowell SF, Mandell LA, File Jr TM, Musher DM, Fine MJ. Practice guidelines for the management of community-acquired pneumonia in adults. Infectious Diseases Society of America. Clin Infect Dis. 2000;31(2):-347-82.
2. Mendes C, Marin ME, Quiñones F, Sifuentes-Osornio J, Siller CC, Castanheira M., et al. Antibacterial resistance of community-acquired respiratory tract pathogens recovered from patients in Latin America: results from the PROTEKT surveillance study (1999-2000). Braz J Infect Dis. 2003;7(1):44-61.
3. Mason EO Jr, Wald ER, Bradley JS, Barson WM, Kaplan SL; United States Pediatric Multicenter Pneumococcal Surveillance Study Group. Macrolide resistance among middle ear isolates of Streptococcus pneumoniae observed at eight United States pediatric centers: prevalence of M and MLSB phenotypes. Pediatr Infect Dis J. 2003;22(7):623-8.
4. Farrel DJ, Jenkins SG. Distribuition across the USA of macrolide resistance and macrolide resistance mechanisms among Streptococcus pneumoniae isolates collected from patients with respiratory tract infections; PROTEKT US 2001-2002. J Antimicrob Chemother 2004;54(Suppl 1):i17-22.
5. Leclercq R, Courvalin P. Resistance to macrolide and related antibiotics in Streptococcus pneumoniae. Antimicrob Agents Chemother. 2002;46(9):2727-34.
6. Montanari MP, Mingoia M, Cochetti H, Varaldo PE. Phenotypes and genotypes of erythromycin-resistant pneumococci in Italy. J Clin Microbiol. 2003;41(1):428-31.
7. Amezaga MR, Carter PE, Cash P, McKenzie H. Molecular epidemiology of erythromycin in Streptococcus pneumoniae isolates from blood and noninvasive sites. J Clin Microbiol. 2002;40(9):3313-8.
8. Song JH, Chang HH, Suh JY, Ko KS, Jung SI, Oh WS, et al. Macrolide resistance and genotypic characterization of Streptococcus pneumoniae in Asian countries: a study of the Asian Network for Surveillance of Resistant Pathogens (ANSORP). J Antimicrob Chemother. 2004; 53(3):457-63.
9. Reinert RR, Franken C, Van der Linden M, Lutticken R, Cil M, Al-Lahham A. Molecular characterization of macrolide resistance mechanisms of Streptococcus pneumoniae and Streptococcus pyogenes isolated in Germany, 2002-2003. Int J Antimicrob Agents. 2004;24(1):43-7.
10. Ruoff KH, Whiley RA, Beighton D. Streptococcus. In: Murray PR, Baron EJ, Pfaller MA, editors. Manual of clinical microbiology. 7a ed. Washington, DC: ASM Press; 1999. p. 283-96.
11. National Committee for Clinical Laboratory Standards. Performance standars for antimicrobial susceptibility testing; Fourteenth International Supplement. Wayne, Pennsylvania; NCCLS; 2004. (Document M100-S14).
12. Sutcliffe J, Tait-Kamradt A, Wondrack L. Streptococcus pneumoniae and Streptococcus pyogenes resistant to macrolide but sensitive to clindamycin: a common resistance pattern mediated by an efflux system. Antimicrob Agents Chemother. 1996;40(8):1817-24.
13. Dias CA. Susceptibilidade a antimicrobianos e diversidade fenotípica e genotípica de Streptococcus pneumoniae isolados em Porto Alegre, Rio Grande do Sul. [tese]. Rio de Janeiro: Universidade Federal do Rio de Janeiro; 2000.
14. Mendonça-Souza CR, Carvalho MG, Barros RR, Dias CA, Sampaio JL, Castro AC, et al. Occurrence and characteristics of erythromycin-resistant Streptococcus pneumoniae strains isolated in three major Brazilian states. Microb Drug Resist. 2004;10(4):313-20.
15. Mendes C, Kiffer CR, Blosser-Middleton RS, Jones ME, Karlowsky JA, Barth A, et al. Antimicrobial susceptibility to levofloxacin and other antibacterial agents among common respiratory pathogens - a Brazilian perspective from the GLOBAL Surveillance Initiative 2001-2002. Clin Microbiol Infect. 2004;10(6):521-6.
16. Bean DC, Klena JD. Prevalence of erm(B) and mef(A) erythromycin resistance determinants in isolates of Streptococcus pneumoniae from New Zealand. J Antimicrob Chemother. 2002;50(4):597-9.
17. Sener B, Koseoglu O. Comparative in vitro activity of antiribosomal agents on penicillin-susceptible and resistant Streptococcus pneumoniae in relation to their resistance genotypes. Int J Antimicrob Agents. 2004;24(1):39-42.
18. Corso A, Severina EP, Petruk VF, Mauriz YR, Tomasz A. Molecular characterization of penicillin-resistant Streptococcus pneumoniae isolates causing respiratory disease in Unites States. Microb Drug Resist. 1998;4(4):325-37.
19. McGee L, Klugman KP, Wasas A, Capper T, Brink A. Serotype 19F multiresistant pneumococcal clone harboring two erythromycin resistance determinants (erm(B) and mef(E)) in South Africa. Antimicrob Agents Chemother. 2001;45(5):1595-8.
20. Farrel DJ, Morrissey I, Bakker S, Buckridge S, Felmingham D. Molecular epidemiology of multiresitant Streptococcus pneumoniae with both ermB- and mef(A)-mediated macrolide resistance. J Clin Microbiol. 2004;42(2):764-8.
* Trabalho realizado no Laboratório de Biologia Molecular - Instituto de Pesquisas Biomédicas da Pontificia Universidade Católica do Rio Grande do Sul - PUCRS - Porto Alegre (RS) Brasil.
1. Mestre em Clínica Médica pela Pontificia Universidade Católica do Rio Grande do Sul - PUCRS - Porto Alegre (RS) Brasil.
2. Doutor em Pneumologia pela Universidade Federal do Rio Grande do Sul - UFRGS - Porto Alegre (RS) Brasil.
3. Pós-Doutorado em Virologia Molecular pela University of Reading, Inglaterra.
4. Graduando em Farmácia pela Pontificia Universidade Católica do Rio Grande do Sul - PUCRS - Porto Alegre (RS) Brasil.
5. Mestre em Microbiologia pela Universidade Federal do Rio de Janeiro - UFRJ - Rio de Janeiro (RJ) Brasil.
6. Doutor em Pneumologia pela Universidade Federal do Rio Grande do Sul - UFRGS - Porto Alegre (RS) Brasil.
Endereço para correspondência: Fabiana Rowe Zettler. Rua General Ibá Mesquita - Ilha Moreira, 180/1401,
Porto Alegre - RS. CEP: 91340-190. Tel.: 55 51 3029-1201. E-mail: ezettler@pucrs.br
Recebido para publicação em 17/2/05. Aprovado, após revisão, em 15/4/05.