ABSTRACT
Obstructive sleep apnea (OSA) has been associated with oxidative stress and various cardiovascular consequences, such as increased cardiovascular disease risk. Quantitative real-time PCR is frequently employed to assess changes in gene expression in experimental models. In this study, we analyzed the effects of chronic intermittent hypoxia (an experimental model of OSA) on housekeeping gene expression in the left cardiac ventricle of rats. Analyses via four different approaches-use of the geNorm, BestKeeper, and NormFinder algorithms; and 2−ΔCt (threshold cycle) data analysis-produced similar results: all genes were found to be suitable for use, glyceraldehyde-3-phosphate dehydrogenase and 18S being classified as the most and the least stable, respectively. The use of more than one housekeeping gene is strongly advised.
Keywords:
Cell hypoxia; Reference standards; Sleep apnea, obstructive; Cardiovascular diseases; Models, animal; Polymerase chain reaction.
RESUMO
A apneia obstrutiva do sono (AOS) tem sido associada ao estresse oxidativo e a várias consequências cardiovasculares, tais como risco aumentado de doença cardiovascular. A PCR quantitativa em tempo real é frequentemente empregada para avaliar alterações na expressão gênica em modelos experimentais. Neste estudo, analisamos os efeitos da hipóxia intermitente crônica (um modelo experimental de AOS) na expressão de genes de referência no ventrículo cardíaco esquerdo de ratos. Análises a partir de quatro abordagens - uso dos algoritmos geNorm, BestKeeper e NormFinder e análise de dados 2−ΔCt (ciclo limiar) - produziram resultados semelhantes: todos os genes mostraram-se adequados para uso, sendo que gliceraldeído-3-fosfato desidrogenase e 18S foram classificados como o mais e o menos estável, respectivamente. A utilização de mais de um gene de referência é altamente recomendada.
Palavras-chave:
Hipóxia celular; Padrões de referência; Apneia do sono tipo obstrutiva; Doenças cardiovasculares; Modelos animais; Reação em cadeia da polimerase.
A apneia obstrutiva do sono (AOS), um distúrbio respiratório do sono cuja prevalência é elevada em todo o mun-do,(1,2) causa diversos distúrbios metabólicos, oxidativos e simpáticos e aumenta o risco de doença cardiovascular.(3) O modelo de hipóxia intermitente crônica (HIC) é o modelo animal de AOS mais usado, especialmente quando o estudo se concentra nas consequências cardiovasculares do distúrbio.(3) O modelo de HIC simula muitos dos efeitos patológicos putativos da AOS, como o aumento da pressão arterial e da atividade simpática.(4)
A PCR em tempo real é a ferramenta mais usada para estudar a expressão gênica de alvos específicos. Para que os resultados da PCR quantitativa em tempo real sejam confiáveis, é necessário usar genes de referência apropria-dos - também denominados housekeeping genes (HKGs)(5) - como controles internos para a normalização da ex-pressão gênica, que é essencial para a avaliação da expressão gênica relativa. No entanto, o uso de certos genes instáveis como HKGs pode comprometer muito a confiabilidade dos dados.(6,7) Portanto, cada condição experimental deveria ter uma etapa de seleção de HKGs para que se obtenham dados de boa qualidade. Os três algoritmos mais usados para a seleção de HKGs são os fornecidos pelos programas de computador geNorm,(8) BestKeeper(9) e Norm-Finder.(10)
Até onde sabemos, não houve estudos que avaliassem a estabilidade da expressão de HKGs no ventrículo esquer-do ou qualquer outra estrutura cardíaca por meio de modelos in vivo de hipóxia; a maioria dos estudos desse tipo foi realizada in vitro. Portanto, o objetivo do presente estudo foi relatar a seleção de HKGs no ventrículo cardíaco es-querdo de ratos submetidos à HIC; os algoritmos do geNorm, BestKeeper e NormFinder foram usados para analisar a estabilidade da expressão de cinco HKGs comumente usados. Nossos resultados fornecem informações importantes para a seleção de HKGs em futuros estudos em que se empreguem modelos de HIC.
Em 22 ratos Wistar Hannover machos adultos, a HIC (8 h/dia) foi induzida conforme se descreveu anteriormente. (11) Todos os procedimentos experimentais foram realizados de acordo com as atuais diretrizes éticas e práticas de uso de animais de laboratório.(12) Este estudo foi aprovado pelo Comitê de Ética em Pesquisa Animal da Universidade Federal de São Paulo (Protocolo n. 2025/11).
Os animais foram divididos em três grupos experimentais: controle (n = 6); HIC (seis semanas de HIC, n = 7) e HIC+N (seis semanas de HIC, seguidas de duas semanas de recuperação em normóxia, n = 8). O protocolo de HIC já foi minuciosamente descrito,(11) assim como o raciocínio por trás do uso de um período de duas semanas de recu-peração no grupo HIC+N.(13)
Após seis semanas (o fim do protocolo de HIC) ou oito semanas (o fim do protocolo de HIC+N), os animais foram eutanasiados por meio de decapitação rápida. Subsequentemente, o coração foi rapidamente removido e lavado com solução salina e, em seguida, o ventrículo esquerdo foi dissecado. Todos os tecidos foram rapidamente disseca-dos em gelo seco e armazenados a −80°C até a extração.
O RNA total foi extraído por meio do reagente TRIzol (Thermo Fisher Scientific Inc., Waltham, MA, EUA) e tratado com DNase. A PCR quantitativa em tempo real foi realizada com o agente SYBR Green, conforme já se descreveu.(11) Para nossa avaliação de HKGs, escolhemos cinco dos genes mais usados, provenientes de diferentes vias genéticas: RNA ribossômico (RNAr) eucariótico 18S; beta-actina (ACTB); beta-2-microglobulina (β2M); gliceraldeído-3-fosfato desidrogenase (GAPDH) e hipoxantina-guanina fosforibosiltransferase (HPRT). As sequências de iniciadores para todos esses HKGs já foram descritas.(11)
A estabilidade da expressão gênica foi avaliada por meio dos programas geNorm (Gene Quantification: http://download.gene-quantification.info/), NormFinder (Department of Molecular Medicine, Aarhus University Hospi-tal, Aarhus, Dinamarca: http://moma.dk/normfinder-software) e BestKeeper (Gene Quantification: http://download.gene-quantification.info/). O programa geNorm calcula a média da estabilidade da expressão (o valor de M) de cada gene; valores de M mais baixos indicam maior estabilidade.(8) O programa NormFinder fornece um valor de estabilidade para cada gene; valores de estabilidade mais baixos indicam menor estabilidade.(10) O programa BestKeeper calcula o coeficiente de correlação de Pearson de cada gene; valores de p mais próximos de 1,0 indicam maior estabilidade.(9) Analisamos também todos os dados de 2−ΔCt (ciclo limiar) por meio de ANOVA de uma via. (14) Para testar a normalidade e a homogeneidade, foram usados o teste de Kolmogorov-Smirnov e o teste de Levene, respectivamente. O nível de significância estatística foi de p < 0,05.
Nossa avaliação da qualidade e integridade do RNA revelou RNAr 18S e 28S intactos. A otimização das condições dos iniciadores e das concentrações de cDNA foi realizada e descrita anteriormente.(11) Os valores de 2−ΔCt, analisa-dos de maneira independente, não atingiram significância (p > 0,05 para todos), embora a expressão de 18S tenha tendido para a significância (p = 0,076 vs. p > 0,4 para os demais genes; dados não apresentados). Esses dados indicam que, embora todos os cinco candidatos a HKG possam ser usados, a expressão de 18S tende a apresentar maior variabilidade entre grupos.
Em nossa análise com o programa BestKeeper para identificar os melhores HKGs em modelos de HIC, todos os cinco genes candidatos apresentaram valores aceitáveis (p < 0,01). Segundo a classificação do BestKeeper, GAPDH foi o melhor candidato, seguido de ACTB. Além disso, 18S apresentou o menor coeficiente de correlação de Pearson, o que significa que foi o menos estável dos cinco candidatos a HKG. Todos os coeficientes de correlação de Pearson do programa BestKeeper encontram-se descritos na Tabela 1 e ilustrados na Figura 1A.

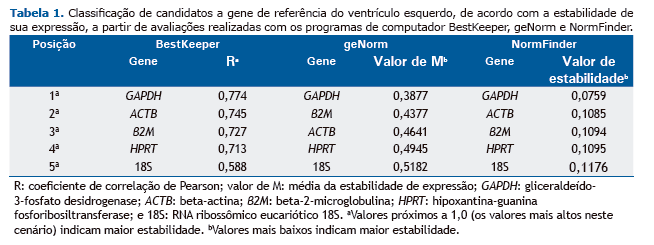
Em nossa análise com o programa geNorm, todos os cinco genes candidatos apresentaram, em todas as análises, valores de M inferiores a 1,5, que é considerado o valor de corte para a adequação.(8) De acordo com a classificação do geNorm, GAPDH foi o melhor gene candidato, seguido de β2M. Além disso, 18S apresentou o maior valor de M e foi considerado o gene menos estável. Todos os valores de M do geNorm encontram-se descritos na Tabela 1 e ilustrados na Figura 1B. Os algoritmos do BestKeeper e do geNorm produziram resultados muito semelhantes; a única diferença foi quanto ao segundo melhor candidato a HKG.
Em nossa análise com o programa NormFinder, todos os cinco genes candidatos apresentaram valores de estabili-dade inferiores a 0,15, o valor de corte para a adequação.(10) Os resultados do NormFinder foram semelhantes, po-rém não idênticos aos do BestKeeper; o algoritmo do NormFinder identificou GAPDH como sendo o melhor gene candidato, seguido de β2M. Além disso, 18S apresentou o maior valor de estabilidade e, portanto, foi considerado o gene menos estável. Todos os valores de estabilidade do NormFinder encontram-se descritos na Tabela 1 e ilustra-dos na Figura 1C.
Foi idêntica a classificação dos HKGs nas avaliações com os programas NormFinder e BestKeeper; GAPDH e ACTB foram os genes mais estáveis, ao passo que 18S foi o menos estável. No entanto, a classificação dos HKGs na avali-ação realizada com o programa geNorm foi ligeiramente diferente: ACTB na terceira posição e β2M na segunda posição. Os dados de todos os três algoritmos corroboram os relatados em um estudo anterior que envolveu células derivadas de cardioesferas (células-tronco cardíacas endógenas, possíveis restauradoras de tecido perdido) e que avaliou a influência do envelhecimento sobre a estabilidade genética. Os autores constataram que ACTB, GAPDH e β2M foram os HKGs mais estáveis em células derivadas de cardioesferas adultas ou neonatais em condições de normóxia ou hipóxia.(16) Embora nosso estudo tenha envolvido a porção do ventrículo esquerdo do coração, que é composta por vários tipos celulares, a origem do tecido e das células derivadas de cardioesferas analisadas por aqueles autores é a mesma. Por outro lado, em um estudo em que se empregou um modelo animal de disfunção diastólica do ventrículo esquerdo, GAPDH foi o segundo gene mais estável, o que ressalta sua estabilidade in vi-vo.(16) Além disso, o gene ACTB apresenta boa estabilidade em modelos que empregam diferentes estímulos de hipóxia, tais como diversos tipos celulares submetidos à hipóxia in vitro(7,15) e diversas estruturas cerebrais subme-tidas à hipóxia in vivo.(11)
Embora análises tenham mostrado que β2M é instável em algumas estruturas cerebrais e linhagens celulares,(7,11) em nossa análise de tecido proveniente do ventrículo esquerdo a β2M ficou entre os genes candidatos mais estáveis. Nossos dados reforçam os de vários outros estudos, nos quais β2M foi um dos genes candidatos mais estáveis em células derivadas de cardioesferas em condições de hipóxia(15) e em condrócitos humanos cultivados com concentra-ções de oxigênio de 5% e 1%.(17)
No presente estudo, HPRT esteve entre os genes candidatos menos estáveis. No entanto, ele pode ser considerado adequado para uso porque apresentou estabilidade aceitável nas análises com o geNorm e o BestKeeper. Nossos dados vão de encontro aos de alguns estudos anteriores, nos quais se relatou que HPRT é um dos genes mais está-veis, em culturas de células submetidas à hipóxia aguda(7) e em um modelo in vivo de isquemia no ventrículo direi-to.(18) No entanto, nossos dados corroboram os de outro estudo anterior com células derivadas de cardioesferas, em que o HPRT esteve entre os genes menos estáveis em condições de hipóxia.(19) É possível que as discrepâncias entre esses estudos se devam a diferenças na origem do tecido avaliado (tecido cardíaco vs. tecido de próstata, rim e mama) e na condição experimental (hipóxia vs. isquemia).
Nossos dados corroboram os de vários outros estudos nos quais 18S foi considerado um HKG inadequado. Em es-tudos em que se empregaram modelos in vitro de hipóxia, o 18S foi considerado inadequado como HKG na linhagem celular LNCaP (de câncer de próstata) (19) e em células musculares de gafanhotos,(6) assim como o observado em diversas estruturas cerebrais um modelo de HIC in vivo.(11) Nossos dados corroboram os de estudos anteriores que demonstraram a sensibilidade do 18S a diferentes estímulos de hipóxia. Até onde sabemos, este é o primeiro estudo a avaliar a expressão de 18S em tecido cardíaco em condições de hipóxia.
Nosso estudo tem algumas limitações. O modelo de HIC simula apenas um dos quatro principais fatores da AOS. Além disso, análises da estabilidade da expressão de HKGs podem demonstrar variabilidade entre modelos e mes-mo entre estruturas.(7,11) Portanto, os resultados de estudos da expressão de HKGs não podem ser generalizados a todas as estruturas ou a todos os modelos de hipóxia. Não obstante, os resultados do presente estudo demonstram que todos os HKGs testados são adequados para uso, embora não seja aconselhável usar o 18S. Além disso, os algoritmos dos programas geNorm, BestKeeper e NormFinder produziram resultados muito robustos, com pouca variação entre os três. No entanto, para que se obtenham dados confiáveis, recomenda-se veementemente o uso de mais de um HKG.
REFERÊNCIAS1. Peppard PE, Young T, Barnet JH, Palta M, Hagen EW, Hla KM. Increased prevalence of sleep-disordered breathing in adults. Am J Epidemiol. 2013;177(9):1006-14. http://dx.doi.org/10.1093/aje/kws342
2. Tufik S, Santos-Silva R, Taddei JA, Bittencourt LR. Obstructive sleep apnea syndrome in the Sao Paulo Epidemiologic Sleep Study. Sleep Med. 2010;11(5):441-6. http://dx.doi.org/10.1016/j.sleep.2009.10.005
3. Dematteis M, Godin-Ribuot D, Arnaud C, Ribuot C, Stanke-Labesque F, Pépin JL, et al. Cardiovascular consequences of sleep-disordered breathing: contribution of animal models to understanding of the human disease. ILAR J. 2009;50(3):262-81. http://dx.doi.org/10.1093/ilar.50.3.262
4. Zoccal DB, Bonagamba LG, Oliveira FR, Antunes-Rodrigues J, Machado BH. Increased sympathetic activity in rats submitted to chronic intermittent hypoxia. Exp Physiol. 2007;92(1):79-85. http://dx.doi.org/10.1113/expphysiol.2006.035501
5. Thellin O, Zorzi W, Lakaye B, De Borman B, Coumans B, Hennen G, et al. Housekeeping genes as internal standards: use and limits. J Biotechnol. 1999;75(2-3):291-5. http://dx.doi.org/10.1016/S0168-1656(99)00163-7
6. Zhao DJ, Guo K, Kang L. Identification of condition-specific reference genes from microarray data for locusts exposed to hypobaric hypoxia. FEBS Open Bio. 2012;2:235-40. http://dx.doi.org/10.1016/j.fob.2012.08.001
7. Caradec J, Sirab N, Keumeugni C, Moutereau S, Chimingqi M, Matar C, et al. "Desperate house genes": the dramatic example of hypoxia. Br J Cancer. 2010;102(6):1037-43. http://dx.doi.org/10.1038/sj.bjc.6605573
8. Vandesompele J, De Preter K, Pattyn F, Poppe B, Van Roy N, De Paepe A, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Genome Biol. 2002;3(7):RESEARCH0034.
9. Pfaffl MW, Tichopad A, Prgomet C, Neuvians TP. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper--Excel-based tool using pair-wise correlations. Biotechnol Lett. 2004;26(6):509-15. http://dx.doi.org/10.1023/B:BILE.0000019559.84305.47
10. Andersen CL, Jensen JL, Ørntoft TF. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets. Cancer Res. 2004;64(15):5245-50. http://dx.doi.org/10.1158/0008-5472.CAN-04-0496
11. Julian GS, de Oliveira RW, Perry JC, Tufik S, Chagas JR. Validation of Housekeeping genes in the brains of rats submitted to chronic intermittent hypoxia, a sleep apnea model. PLoS One. 2014;9(10):e109902. http://dx.doi.org/10.1371/journal.pone.0109902
12. Andersen M, Tufik S. Animal models as ethical tools in biomedical research. São Paulo: Universidade Federal de São Paulo; 2010. 563 pp.
13. Veasey SC, Davis CW, Fenik P, Zhan G, Hsu YJ, Pratico D, et al. Long-term intermittent hypoxia in mice: protracted hypersomnolence with oxidative injury to sleep-wake brain regions. Sleep. 2004;27(2):194-201.
14. Silver N, Best S, Jiang J, Thein SL. Selection of housekeeping genes for gene expression studies in human reticulocytes using real-time PCR. BMC Mol Biol. 2006;7:33. http://dx.doi.org/10.1186/1471-2199-7-33
15. Tan SC, Carr CA, Yeoh KK, Schofield CJ, Davies KE, Clarke K. Identification of valid housekeeping genes for quantitative RT-PCR analysis of cardiosphere-derived cells preconditioned under hypoxia or with prolyl-4-hydroxylase inhibitors. Mol Biol Rep. 2012;39(4):4857-67. http://dx.doi.org/10.1007/s11033-011-1281-5
16. Nachar W, Busseuil D, Shi Y, Mihalache-Avram T, Mecteau M, Rhéaume E, et al. Optimisation of reference genes for gene-expression analysis in a rabbit model of left ventricular diastolic dysfunction. PLoS One. 2014;9(2):e89331. http://dx.doi.org/10.1371/journal.pone.0089331
17. Foldager CB, Munir S, Ulrik-Vinther M, Søballe K, Bünger C, Lind M. Validation of suitable house keeping genes for hypoxia-cultured human chondrocytes. BMC Mol Biol. 2009;10:94. http://dx.doi.org/10.1186/1471-2199-10-94
18. Vesentini N, Barsanti C, Martino A, Kusmic C, Ripoli A, Rossi A, et al. Selection of reference genes in different myocardial regions of an in vivo ischemia/reperfusion rat model for normalization of antioxidant gene expression. BMC Res Notes. 2012;5:124. http://dx.doi.org/10.1186/1756-0500-5-124
19. Yao L, Chen X, Tian Y, Lu H, Zhang P, Shi Q, et al. Selection of housekeeping genes for normalization of RT-PCR in hypoxic neural stem cells of rat in vitro. Mol Biol Rep. 2012;39(1):569-76. http://dx.doi.org/10.1007/s11033-011-0772-8
20. Caradec J, Sirab N, Keumeugni C, Revaud D, Loric S. Reply: 18S is an appropriate housekeeping gene for in vitro hypoxia experiments. Br J Cancer. 2010;103(4):591-2. http://dx.doi.org/10.1038/sj.bjc.6605755
21. Nagelkerke A, Mujcic H, Wouters B, Span PN. 18S is an appropriate housekeeping gene for in vitro hypoxia experiments. Br J Cancer. 2010;103(4):590; author reply 591-2. http://dx.doi.org/10.1038/sj.bjc.6605754