ABSTRACT
Objective: To determine whether polymorphisms of the IL10 and IL17 genes are associated with severe asthma control and bronchodilator reversibility in children and adolescents with severe asthma. Methods: This was a cross-sectional study, nested within a prospective cohort study of patients with severe asthma. Two outcomes were evaluated: asthma control and bronchodilator reversibility. We extracted DNA from peripheral blood and genotyped three single nucleotide polymorphisms: rs3819024 and rs2275913 in the IL17A gene; and rs3024498 in the IL10 gene. For the association analyses, we performed logistic regression in three genetic models (allelic, additive, and dominant). Results: The rs3024498 C allele in the IL10 gene was associated with failure to achieve asthma control despite regular treatment (p = 0.02). However, the G allele of the IL17A rs3819024 polymorphism was associated with failure to respond to stimulation with a ß2 agonist. The rs2275913 polymorphism of the IL17A gene showed no relationship with asthma control or bronchodilator reversibility. Conclusions: In pediatric patients with severe asthma, the IL10 polymorphism appears to be associated with failure to achieve clinical control, whereas the IL17A polymorphism appears to be associated with a worse bronchodilator response. Knowledge of the involvement of these polymorphisms opens future directions for pharmacogenetic studies and for the implementation of individualized therapeutic management of severe asthma in pediatric patients.
Keywords:
Polymorphism, genetic; Interleukin-10; Interleukin-17; Asthma.
RESUMO
Objetivo: Determinar se existe relação entre polimorfismos dos genes IL10 e IL17 e controle da asma grave e reversibilidade com broncodilatador em crianças e adolescentes com asma grave. Métodos: Estudo transversal, aninhado em um estudo prospectivo de coorte com pacientes com asma grave. Foram avaliados dois desfechos: controle da asma e reversibilidade com broncodilatador. Extraímos DNA do sangue periférico e genotipamos três polimorfismos de nucleotídeo único: rs3819024 e rs2275913 no gene IL17A e rs3024498 no gene IL10. Para as análises de associação, realizamos regressão logística em três modelos genéticos (alélico, aditivo e dominante). Resultados: O alelo C do polimorfismo rs3024498 do gene IL10 apresentou relação com asma que permaneceu descontrolada mesmo com tratamento regular (p = 0,02). No entanto, o alelo G do polimorfismo rs3819024 do gene IL17A apresentou relação com ausência de resposta ao estímulo com β2-agonista. O polimorfismo rs2275913 do gene IL17A não apresentou relação com controle da asma ou reversibilidade com broncodilatador. Conclusões: Em pacientes pediátricos com asma grave, o polimorfismo do gene IL10 parece estar relacionado com ausência de controle clínico, ao passo que o polimorfismo do gene IL17A parece estar relacionado com pior resposta ao broncodilatador. O conhecimento a respeito do envolvimento desses polimorfismos abre perspectivas futuras para estudos farmacogenéticos e para a implantação de manejo terapêutico individualizado da asma grave em pacientes pediátricos.
Palavras-chave:
Polimorfismo genético; Interleucina-10; Interleucina-17; Asma.
INTRODUÇÃO Embora afete apenas aproximadamente 2% de todas as crianças com asma, a asma grave está associada a uma alta taxa de morbidade.(1,2) A forma refratária da asma grave é caracterizada por mau controle contínuo, mesmo com o uso de doses máximas de medicamentos de controle e a abordagem de fatores modificáveis como adesão ao tratamento, exposição a alérgenos e exposição ao tabagismo. Um dos principais motivos da falta de controle é provavelmente a natureza heterogênea da doença, cuja patogênese envolve a interação de fatores ambientais e variabilidade individual, além de uma base genética complexa.(3)
A heterogeneidade da asma grave pode ser explicada por fenótipos moleculares distintos com citocinas não relacionadas à via clássica dos linfócitos Th2, tais como IL-10 e IL-17.(4) Ao inibir a produção de citocinas pró-inflamatórias, a IL-10 reduz a inflamação alérgica; o mau controle clínico em pacientes com asma grave provavelmente está relacionado à ausência de resposta a doses crescentes de esteroides inalatórios.(5) Por sua vez, a IL-17 estimula a produção de citocinas inflamatórias Th17, promovendo assim a inflamação das vias aéreas.(6)
Polimorfismos dos genes IL10 e IL17 foram descritos em uma meta-análise de pacientes pediátricos de diferentes etnias, embora apenas o risco de asma tenha sido avaliado. (6,7) Polimorfismos desses genes de citocinas poderiam explicar a dificuldade em alcançar o controle clínico e funcional em pacientes com asma grave. No entanto, até onde sabemos, nenhum estudo estabeleceu uma relação entre variantes dos genes IL10 e IL17 e controle clínico em casos de asma grave. Como polimorfismos desses genes de citocinas podem ser biomarcadores úteis para ajustar esquemas terapêuticos, o objetivo do presente estudo foi avaliar se polimorfismos dos genes IL10 e IL17 apresentavam relação com o controle da asma grave e a resposta ao broncodilatador em uma amostra composta por pacientes pediátricos.
MÉTODOS Desenho do estudo, participantes e características clínicas Trata-se de um estudo transversal, aninhado em um estudo prospectivo de coorte com pacientes com asma grave,(8) realizado entre 2021 e 2022 no Centro Multidisciplinar de Asma de Difícil Controle e no Instituto de Ciências Biológicas da Universidade Federal de Minas Gerais, em Belo Horizonte (MG). O estudo foi realizado em colaboração com o Laboratório de Imunofarmacologia e Biologia Molecular do Instituto de Ciências da Saúde da Universidade Federal da Bahia, em Salvador (BA). Selecionamos pacientes que apresentavam asma grave (cuja definição foi asma confirmada por meio de medida objetiva da função pulmonar), que apresentavam boa adesão ao tratamento e que, apesar da eliminação ou minimização de fatores associados ao mau controle da doença, necessitavam de altas doses de corticosteroides inalatórios (CI — budesonida ≥ 1.600 µg ou equivalente) e um segundo medicamento de controle — long-acting β2 agonists (LABAs, β2-agonistas de longa duração), antagonistas muscarínicos de longa duração, antileucotrienos ou qualquer combinação dos três — ou corticosteroides orais ao longo de 50% ou mais do ano anterior para manter o controle da doença, bem como aqueles nos quais a doença permaneceu descontrolada em virtude de sua gravidade intrínseca.(9-11) Como a asma grave refratária é um fenótipo incomum, a amostragem foi de conveniência. Realizamos o cálculo do tamanho da amostra a posteriori, por meio do programa OpenEpi, versão 3.01 (um programa gratuito de código aberto, disponível on-line), e constatamos que, para obter um poder estatístico de 80%, seria necessária uma amostra composta por 34 indivíduos em cada grupo.(12)
Foram excluídos pacientes que eram parentes de primeiro grau (irmãos, por exemplo), bem como aqueles com outras doenças pulmonares crônicas. Indivíduos saudáveis não foram incluídos no presente estudo.
Foram avaliadas características clínicas como idade (em anos), idade no início do tratamento com CI (em meses), tabagismo materno durante a gestação, relato de tabagismo passivo, admissão prévia na UTI em virtude de asma, exacerbações graves nos últimos 12 meses, peso (em kg), estatura (em cm) e IMC (em kg/m2).
A dose de CI foi avaliada quanto a sua equivalência à da budesonida. A taxa de adesão ao tratamento com CI foi considerada ótima quando esteve acima de 80%. Ela foi calculada como a proporção da dose total recomendada, por meio da verificação do contador de doses dos inaladores dosimetrados pressurizados ou da contagem das cápsulas usadas nos inaladores de pó.(9) Alguns pacientes usavam inaladores de pó com associação medicamentosa de budesonida e formoterol (Alenia; Aché Laboratórios Farmacêuticos S/A, Guarulhos, Brasil), ao passo que outros usavam inaladores dosimetrados pressurizados com associação medicamentosa de fluticasona e salmeterol (Seretide; GlaxoSmithKline, Stevenage, Reino Unido) ou omalizumabe apenas (Xolair; Novartis Biociências S/A., São Paulo, Brasil). O uso de antileucotrienos e imunobiológicos também foi avaliado.
O diagnóstico de rinite alérgica baseou-se na história clínica do paciente, em um questionário de sintomas nasais e no resultado positivo para aeroalérgenos no teste cutâneo.(9) Além da rinite, foram consideradas outras comorbidades: dermatite atópica, respiração bucal, doença do refluxo gastroesofágico, distúrbios comportamentais e distúrbios emocionais.(10)
Procedimentos O nível de controle da asma foi avaliado por meio da aplicação dos critérios da GINA.(10) Os pacientes responderam se nas últimas quatro semanas haviam apresentado sintomas de asma durante o dia, mais de duas vezes por semana; acordado à noite por causa da asma; usado um short-acting β2 agonist (SABA, β2-agonista de curta duração) para aliviar os sintomas da asma mais de duas vezes por semana; e experimentado alguma limitação de atividades por causa da asma. A definição de asma controlada foi uma resposta negativa a todas as quatro perguntas, ao passo que a definição de asma não controlada foi uma resposta afirmativa a qualquer uma das quatro perguntas. Com base em suas respostas, os pacientes foram divididos em dois grupos: asma grave controlada e asma grave não controlada.
Testes cutâneos por punctura foram realizados e foram considerados positivos nos casos em que a pápula foi pelo menos 3 mm maior que a do controle negativo.(13) Testamos os seguintes alérgenos, todos obtidos junto ao mesmo fornecedor (Imunotec, São Paulo, Brasil): Dermatophagoides pteronyssinus; Dermatophagoides farinae; Blomia tropicalis; epitélio de cão e gato; Aspergillus sp.; Penicillium sp.; Periplaneta americana; e Cladosporium sp. Também foram medidos os eosinófilos no sangue periférico e os níveis séricos de citocinas. No entanto, estes últimos não foram superiores ao limite inferior de detecção.
Todos os pacientes foram submetidos a testes de função pulmonar, realizados com espirômetro KoKo (KoKo PFT, Longmont, CO, EUA) e em conformidade com as recomendações da American Thoracic Society. (14) O VEF1, a CVF e a relação VEF1/CVF foram avaliados antes e depois da administração de 400 µg de salbutamol por meio de um inalador dosimetrado pressurizado. A definição de variação significativa pós-broncodilatador, ou reversibilidade com broncodilatador, foi um aumento de 200 mL ou 12% no VEF1.(15)
Extração do DNA genômico, genotipagem e análise in silico Amostras de sangue periférico foram coletadas sob vácuo em tubos de 10 mL contendo o anticoagulante ácido etilenodiaminotetracético (Vacutainer; Becton Dickinson, Sparks, MD, EUA) e centrifugadas em centrífuga Kasvi (K14-0815A; Kasvi, São José dos Pinhais, Brasil) a 3.000 rpm durante 10 min a 4°C. O plasma e a camada leucocitária foram separados e então colocados em tubos Eppendorf e armazenados a −30°C. Para a extração do DNA, usamos um kit comercial (Gentra Puregene; QIAGEN, Hilden, Alemanha). Todas as amostras genotipadas foram padronizadas na concentração de 5 ng/µL e armazenadas a −30°C até o uso.
Com base em estudos de associação a respeito da asma,(16-19) selecionamos três polimorfismos de nucleotídeo único (SNPs, do inglês single nucleotide polymorphisms) genotipados sabidamente associados à asma: rs3819024 e rs2275913 no gene IL17A e rs3024498 no gene IL10. A genotipagem foi realizada por meio da tecnologia TaqMan (TaqMan probe-based 5’-nuclease assays; Applied Biosystems, Foster City, CA, EUA) no sistema QuantStudio 12K Flex de reação em cadeia da polimerase em tempo real (Applied Biosystems). Em nossa análise, incluímos apenas os SNPs com call rate (taxa de genotipagem) de no mínimo 93%. Como controles negativos, usamos poços sem DNA para avaliar a amplificação inespecífica.
As informações sobre a função de cada variante de nucleotídeo único foram obtidas no site do National Center for Biotechnology Information, dos EUA (www.ncbi.nlm.nih.gov). Além disso, o banco de dados RegulomeDB foi usado para identificar potenciais variantes regulatórias e funcionais por meio de previsões computacionais e anotações manuais.(20) O banco de dados atribui uma pontuação que vai de 1 a 6; pontuações mais baixas indicam evidências crescentes de que uma variante está localizada em uma região funcional.(20)
HaploReg (Broad Institute, Cambridge, MA, EUA) é uma ferramenta que permite aos pesquisadores explorar anotações do genoma não codificante em variantes em blocos de haplótipos. A ferramenta se concentra especificamente na identificação de candidatas a variantes de nucleotídeo único regulatórias em loci associados a doenças. A HaploReg é uma ferramenta projetada para auxiliar pesquisadores na elaboração de hipóteses mecanicistas a respeito do impacto de variantes não codificantes em fenótipos clínicos e na variação normal.(21)
O Genotype-Tissue Expression Project (Projeto GTEx) dos National Institutes of Health, dos EUA (www.gtexportal.org) tem propiciado insights valiosos a respeito da associação entre expressão gênica, variação genética e outros fenótipos moleculares em diversos tecidos humanos.(22) Por meio de expression quantitative trait loci mapping (mapeamento de loci de características quantitativas de expressão), podemos investigar de modo eficaz os fatores genéticos responsáveis por alterações na expressão gênica. Por meio dessa ferramenta, extraímos informações especificamente relacionadas à influência de variantes do gene de interesse em sua expressão em amostras de sangue total.
Análise estatística A distribuição das variáveis contínuas foi analisada por meio do teste de Shapiro-Wilk. Os dados estão expressos em forma de média ± desvio padrão, mediana [intervalo interquartil] ou frequências absolutas e relativas, dependendo do tipo de variável. Para as comparações entre os grupos (pacientes vs. controles ou genótipo vs. genótipo), usamos o teste t de Student não pareado ou o teste de Mann-Whitney, conforme apropriado. Valores de p < 0,05 foram considerados significativos.
As análises de associação foram realizadas por regressão logística em três modelos genéticos (alélico, aditivo e dominante), ajustados pelas covariáveis sexo e idade, por meio do programa PLINK, versão 1.9.(23) Para reduzir a chance de associações com valores falso-positivos de p,(23) foram consideradas estatisticamente significativas apenas as associações de SNPs com valores de p < 0,05, que foram revalidadas por procedimentos de permutação.
O estudo foi aprovado pelo Comitê de Ética em Pesquisa da Universidade Federal de Minas Gerais (Protocolo n. 4.048.940). Todos os participantes ou responsáveis assinaram um termo de consentimento livre e esclarecido.
RESULTADOS Inicialmente, 62 pacientes com asma grave estavam aptos para participar do estudo. No entanto, 6 foram excluídos porque eram parentes de primeiro grau de outros pacientes selecionados. Portanto, a amostra foi composta por 56 pacientes com asma grave: 19 no grupo com asma controlada e 37 no grupo com asma não controlada. As características demográficas, clínicas e funcionais da população estudada são apresentadas na Tabela 1.
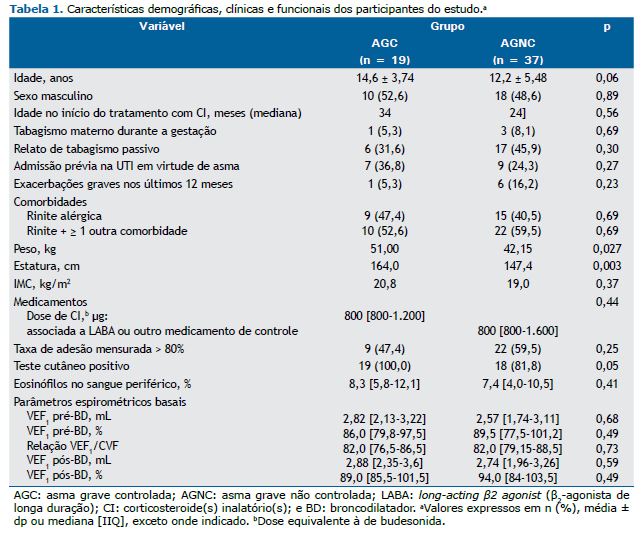
Não observamos nenhuma diferença significativa entre os dois grupos de pacientes com asma grave quanto a variáveis funcionais e marcadores biológicos, o que mostra que esses marcadores não discriminam asma controlada de asma não controlada. Não houve diferença entre os dois grupos quanto ao IMC. No entanto, a média de estatura foi menor no grupo de pacientes com asma não controlada. Ambos os grupos de pacientes usavam altas doses de CI e altas doses de outros medicamentos de controle, e não houve diferença significativa entre os dois grupos quanto às doses ou à adesão ao tratamento.
A Tabela 2 apresenta as características dos SNPs estudados. A frequência alélica menor foi superior a 10% para todos os três SNPs genotipados. Nenhuma variante foi excluída pelo equilíbrio de Hardy-Weinberg ou por call rate baixa. As variantes do gene IL17A estavam numa região não traduzida 2 kb a montante do gene, e a variante do gene IL10 estava na região 3’ não traduzida.
Como se pode observar na Tabela 3, o risco de asma não controlada mesmo com tratamento regular foi maior nos pacientes com pelo menos um alelo C do polimorfismo rs3024498 do gene IL10. Nenhuma das outras duas variantes relacionou-se com falha de controle da asma grave em nenhum dos modelos genéticos testados.
A resposta ao broncodilatador foi menor nos pacientes com asma grave com genótipo AG ou GG de rs3819024 do que naqueles com genótipo AA (Figura 1). Nenhuma das outras duas variantes relacionou-se com ausência de reversibilidade com broncodilatador em nenhum dos modelos genéticos testados. O SNP rs2275913 do gene IL17A não apresentou nenhuma relação com o controle da doença ou a reversibilidade com broncodilatador. Por meio do Projeto GTEx, descobrimos que os genótipos TC e CC de rs3024498 apresentaram maior expressão de IL-10 em amostras de sangue total do que o genótipo TT (Figura 2; p = 0,000053).
DISCUSSÃO Em pacientes pediátricos, a inflamação das vias aéreas relacionada ao processo alérgico é tipicamente eosinofílica e orquestrada por células Th2. No entanto, eosinofilia não é sinônimo de ativação de resposta mediada por células Th2.(4,24) Isso foi demonstrado em um estudo com pacientes pediátricos com asma grave refratária ao tratamento, no qual não foram observados níveis significativos de IL-4, IL-5 ou IL-13 em amostras de escarro ou em espécimes obtidos por meio de biópsia endobrônquica.(25) Portanto, ainda não está claro qual mecanismo imunológico explicaria a conversão da eosinofilia em asma e o fenótipo de gravidade. No presente estudo, demonstramos que polimorfismos dos genes IL10 e IL17 relacionaram-se com asma não controlada e com a resposta ao broncodilatador em pacientes com asma grave. O mau controle da asma relacionou-se com a presença de pelo menos um alelo C do polimorfismo rs3024498 do gene IL10 em pacientes em tratamento regular com altas doses de CI e LABAs ou outros medicamentos de controle. Sabe-se que um dos objetivos do tratamento da asma é o controle dos sintomas com a menor dose possível de medicação inalatória.
Com base nos dados fornecidos pelo Projeto GTEx, indivíduos com pelo menos um alelo C do polimorfismo rs3024498 do gene IL10 apresentam expressão aumentada de IL-10 no sangue. Embora não tenhamos conseguido medir os níveis de IL-10 em nossa população, Rogers et al.(26) observaram níveis mais elevados de IL-10 em crianças com asma não controlada do que naquelas com asma controlada. A forma variante desse gene provavelmente contribui para essa disparidade.(26)
Diante do fato de que doses elevadas de CI têm efeitos adversos importantes, o conhecimento de que a falta de controle da asma pode estar relacionada ao polimorfismo rs3024498 do gene IL10 poderia ser usado para orientar a conduta médica, minimizando assim os riscos associados ao uso contínuo de CI e a aumentos progressivos da dosagem.
Até onde sabemos, não há estudos que relacionem o polimorfismo rs3024498 do gene IL10 com a falha de controle da asma pediátrica. No entanto, há relatos de que esse polimorfismo está relacionado com diversas outras condições clínicas com processo inflamatório exuberante, tais como lúpus eritematoso sistêmico, artrite reumatoide e hepatite crônica.(27,28) Além disso, outros polimorfismos do gene IL10 (rs1800896, rs1800871, rs302109, rs1800872 e rs3024491) foram associados à asma pediátrica.(7,29)
Ao avaliar a resposta ao broncodilatador, observamos que os pacientes com o genótipo homozigoto AA de rs3819024 (IL17A) apresentavam maior probabilidade de resposta ao estímulo agudo de um SABA do que aqueles com o genótipo AG ou GG. Embora tenhamos avaliado a resposta a um SABA, deve-se ter em mente que o formoterol, embora seja um LABA e seja recomendado em todos os cenários de asma grave, tem início de ação semelhante ao do salbutamol.(10) Isso é consistente com os achados de estudos que indicam que a IL-17A atua diretamente na musculatura lisa das vias aéreas, aumentando a contratilidade.(4,30) Na prática clínica, o bloqueio da sinalização de IL17 poderia ser um alvo atraente para o tratamento da asma com fenótipo Th17.(4)
Embora o efeito específico do polimorfismo rs3819024 na expressão de IL-17A permaneça desconhecido, é plausível que essa variante aumente a produção dessa citocina. Esta suposição é apoiada por sua localização 2 kb a montante com alto potencial regulador, conforme indicado pela pontuação = 1f no banco de dados RegulomeDB, com potencial para impactar a interação com histonas nas regiões promotora (H3K4me3) e enhancer (intensificadora; H3K4me1) de linfócitos Th17, conforme descrito na ferramenta HaploReg.
Vale ressaltar que o mau controle dos sintomas nem sempre é atribuível à má adesão ao tratamento com CI e LABAs ou outros medicamentos de controle. Em nossa população, a taxa de adesão ao tratamento foi boa nos grupos de pacientes com asma controlada e não controlada, sem diferença significativa entre os dois. No entanto, a expressão fenotípica depende da interação de fatores ambientais e da predisposição genética de um indivíduo, partindo-se do princípio de que os genes não operam isoladamente, mas sim em seu ambiente (que inclui outros genes ao seu redor), o que pode modificar e até reverter completamente seus efeitos.(3) Portanto, em virtude do alto custo para a sociedade como um todo, estudos que gerem evidências a partir da fenotipagem, endotipagem e genotipagem devem ser incentivados, a fim de identificar os pacientes-alvo e despender recursos de forma mais racional, como tem sido feito em outras doenças crônicas.
Nosso estudo tem algumas limitações. Uma delas é a amostra pequena, que pode ser atribuída ao fato de que a asma grave refratária é um fenótipo incomum. O cálculo do tamanho da amostra foi realizado a posteriori por meio do programa OpenEpi e mostrou que, para os desfechos do estudo, com poder estatístico de 80%, seria necessária uma amostra composta por no mínimo 34 indivíduos por grupo.(12) Nossos pacientes foram selecionados a partir de uma coorte de pacientes que tinham diagnóstico bem estabelecido de asma grave e que estavam em acompanhamento regular em longo prazo, no qual foram usados vários métodos para quantificar a adesão, minimizar a exposição a alérgenos e controlar as comorbidades, limitando assim o tamanho de nossa amostra. Portanto, são necessários estudos com amostras maiores para replicar nossos achados e identificar outros genes que possam explicar a falta de controle da doença em pacientes com asma grave. Estudos futuros também deverão ser realizados com o objetivo de analisar o perfil imunológico, incluindo outras citocinas, o que ajudará a compreender a fisiopatologia complexa e heterogênea da asma grave. Outra limitação do presente estudo é que não foram quantificados os níveis de IL-10 e IL-17. Isso ocorreu porque as amostras passaram por variabilidade térmica, o que poderia ter desnaturado as citocinas.
Em suma, polimorfismos dos genes IL10 e IL17 parecem estar envolvidos em complexas vias de modulação relacionadas à falta de controle da doença e à resposta ao broncodilatador na asma grave refratária ao tratamento em crianças e adolescentes. Estudos funcionais devem ser realizados para caracterizar o impacto molecular dessas variantes, o que poderia facilitar a implantação do tratamento e manejo personalizado da asma.
AGRADECIMENTOS Gostaríamos de agradecer a todos os pacientes que se voluntariaram para participar deste estudo, inclusive seus familiares. Gostaríamos também de expressar nosso agradecimento especial a Diego Menezes, técnico do Laboratório de Genética do Instituto de Ciências Biológicas da Universidade Federal de Minas Gerais, por sua contribuição para o processamento inicial das amostras.
CONTRIBUIÇÕES DOS AUTORES MIRV: investigação, metodologia, administração do projeto e redação do manuscrito original. LMLBFL e MVNPQ: conceituação, curadoria dos dados, análise formal, investigação, metodologia, recursos, supervisão, redação do manuscrito original e revisão e edição do manuscrito. RSC: conceituação, análise formal, investigação, metodologia, recursos, supervisão, redação do manuscrito original e revisão e edição do manuscrito. MBRS, HSS, AO e CAVF: metodologia, análise formal, redação do manuscrito original e revisão e edição do manuscrito. EMTS: curadoria dos dados, investigação, metodologia, análise formal, validação, redação do manuscrito original e revisão e edição do manuscrito.
CONFLITOS DE INTERESSE Nenhum declarado.
REFERÊNCIAS 1.Busse WW. Definition and impact. In: Chung KF, Israel E, Gibson PG. (eds.). Severe asthma (ERS Monograph). Sheffield: European Respiratory Society; 2019; p. 1-15. https://doi.org/10.1183/2312508X.10022418
2.Kuruvilla ME, Lee FE, Lee GB. Understanding Asthma Phenotypes, Endotypes, and Mechanisms of Disease. Clin Rev Allergy Immunol. 2019;56(2):219-233. https://doi.org/10.1007/s12016-018-8712-1
3.Bush A. Genes in their environment: how can we read the riddles?. J Pediatr (Rio J). 2008;84(3):185-188. https://doi.org/10.2223/JPED.1789
4.Ramakrishnan RK, Al Heialy S, Hamid Q. Role of IL-17 in asthma pathogenesis and its implications for the clinic. Expert Rev Respir Med. 2019;13(11):1057-1068. https://doi.org/10.1080/17476348.2019.1666002
5.Gupta A, Dimeloe S, Richards DF, Chambers ES, Black C, Urry Z, et al. Defective IL-10 expression and in vitro steroid-induced IL-17A in paediatric severe therapy-resistant asthma. Thorax. 2014;69(6):508-515. https://doi.org/10.1136/thoraxjnl-2013-203421
6.Wang YH, Wills-Karp M. The potential role of interleukin-17 in severe asthma. Curr Allergy Asthma Rep. 2011;11(5):388-394. https://doi.org/10.1007/s11882-011-0210-y
7.Huang ZY, Cheng BJ, Wan Y, Zhou C. Meta-analysis of the IL-10 promoter polymorphisms and pediatric asthma susceptibility. Genet Mol Res. 2016;15(2):10.4238/gmr.15028320. https://doi.org/10.4238/gmr.15028320
8.Versiani Nunes Pinheiro de Queiroz M, Gonçalves Alvim C, Cruz ÁA, de Lima Belizário Facury Lasmar LM. Lung function in severe pediatric asthma: a longitudinal study in children and adolescents in Brazil. Clin Transl Allergy. 2017;7:48. https://doi.org/10.1186/s13601-017-0183-6
9.Carvalho-Pinto RM, Cançado JED, Pizzichini MMM, Fiterman J, Rubin AS, Cerci Neto A, et al. 2021 Brazilian Thoracic Association recommendations for the management of severe asthma. J Bras Pneumol. 2021;47(6):e20210273. https://doi.org/10.36416/1806-3756/e20210273
10.Global Initiative for Asthma (GINA) [homepage on the Internet]. Bethesda: GINA; c2022 [cited 2022 May 1]. Global Strategy for Asthma Management and Prevention (Updated 2022). [Adobe Acrobat document, 225p.]. Available from: https://ginasthma.org/wp content/uploads/2022/05/GINA Main Report 2022 FINAL 22 05 03 WMS.pdf
11.Chung KF, Wenzel SE, Brozek JL, Bush A, Castro M, Sterk PJ, et al. International ERS/ATS guidelines on definition, evaluation and treatment of severe asthma [published correction appears in Eur Respir J. 2014 Apr;43(4):1216. Dosage error in article text] [published correction appears in Eur Respir J. 2018 Jul 27;52(1):] [published correction appears in Eur Respir J. 2022 Jun 9;59(6):]. Eur Respir J. 2014;43(2):343-373. https://doi.org/10.1183/09031936.00202013
12.Dean AG, Sullivan KM, Soe MM. OpenEpi: Open Source Epidemiologic Statistics for Public Health, Version. www.OpenEpi.com [updated 2013 Apr 6]. Available from: https://www.openepi.com
13.Bousquet J, Heinzerling L, Bachert C, Papadopoulos NG, Bousquet PJ, Burney PG, et al. Practical guide to skin prick tests in allergy to aeroallergens. Allergy. 2012;67(1):18-24. https://doi.org/10.1111/j.1398-9995.2011.02728.x
14.Beydon N, Davis SD, Lombardi E, Allen JL, Arets HGM, Aurora P, et al. An official American Thoracic Society/European Respiratory Society statement: pulmonary function testing in preschool children. Am J Respir Crit Care Med. 2007;175(12):1304-1345. https://doi.org/10.1164/rccm.200605-642ST
15.Graham BL, Steenbruggen I, Miller MR, Barjaktarevic IZ, Cooper BG, Hall GL, et al. Standardization of Spirometry 2019 Update. An Official American Thoracic Society and European Respiratory Society Technical Statement. Am J Respir Crit Care Med. 2019;200(8):e70-e88. https://doi.org/10.1164/rccm.201908-1590ST
16.Holster A, Teräsjärvi J, Lauhkonen E, Törmänen S, Helminen M, Koponen P, et al. IL-17A gene polymorphism rs2275913 is associated with the development of asthma after bronchiolitis in infancy. Allergol Int. 2018;67(1):109-113. https://doi.org/10.1016/j.alit.2017.05.010
17.Maalmi H, Beraies A, Charad R, Ammar J, Hamzaoui K, Hamzaoui A. IL-17A and IL-17F genes variants and susceptibility to childhood asthma in Tunisia. J Asthma. 2014;51(4):348-354. https://doi.org/10.3109/02770903.2013.876647
18.Chen J, Deng Y, Zhao J, Luo Z, Peng W, Yang J, et al. The polymorphism of IL-17 G-152A was associated with childhood asthma and bacterial colonization of the hypopharynx in bronchiolitis. J Clin Immunol. 2010;30(4):539-545. https://doi.org/10.1007/s10875-010-9391-8
19.Figueiredo CA, Barreto ML, Alcantara-Neves NM, Rodrigues LC, Cooper PJ, Cruz AA, et al. Coassociations between IL10 polymorphisms, IL-10 production, helminth infection, and asthma/wheeze in an urban tropical population in Brazil [published correction appears in J Allergy Clin Immunol. 2013 Oct;132(4):1015]. J Allergy Clin Immunol. 2013;131(6):1683-1690. https://doi.org/10.1016/j.jaci.2012.10.043
20.Boyle AP, Hong EL, Hariharan M, Cheng Y, Schaub MA, Kasowski M, et al. Annotation of functional variation in personal genomes using RegulomeDB. Genome Res. 2012;22(9):1790-1797. https://doi.org/10.1101/gr.137323.112
21.Ward LD, Kellis M. HaploReg: a resource for exploring chromatin states, conservation, and regulatory motif alterations within sets of genetically linked variants. Nucleic Acids Res. 2012;40(Database issue):D930-D934. https://doi.org/10.1093/nar/gkr917
22.GTEx Consortium. The Genotype-Tissue Expression (GTEx) project. Nat Genet. 2013;45(6):580-585. https://doi.org/10.1038/ng.2653
23.Purcell S, Neale B, Todd-Brown K, Thomas L, Ferreira MAR, Bender D, et al. PLINK: a tool set for whole-genome association and population-based linkage analyses. Am J Hum Genet. 2007;81(3):559-575. https://doi.org/10.1086/519795
24.Woodruff PG, Modrek B, Choy DF, Jia G, Abbas AR, Ellwanger A, et al. T-helper type 2-driven inflammation defines major subphenotypes of asthma [published correction appears in Am J Respir Crit Care Med. 2009 Oct 15;180(8):796]. Am J Respir Crit Care Med. 2009;180(5):388-395. https://doi.org/10.1164/rccm.200903-0392OC
25.Bossley CJ, Fleming L, Gupta A, Regamey N, Frith J, Oates T, et al. Pediatric severe asthma is characterized by eosinophilia and remodeling without T(H)2 cytokines. J Allergy Clin Immunol. 2012;129(4):974-82.e13. https://doi.org/10.1016/j.jaci.2012.01.059
26.Rogers VE, Bollinger ME, Tulapurkar ME, Zhu S, Hasday JD, Pereira KD, et al. Inflammation and asthma control in children with comorbid obstructive sleep apnea. Pediatr Pulmonol. 2018;53(9):1200-1207. https://doi.org/10.1002/ppul.24074
27.Lv TT, Wu J, Li J, Zhang TP, Yang XK, Xiang N, et al. Association of interleukin-10 gene single nucleotide polymorphisms with susceptibility to systemic lupus erythematosus in a Chinese population. Gene. 2018;642:549-554. https://doi.org/10.1016/j.gene.2017.11.072
28.Świątek-Kościelna B, Kałużna E, Strauss E, Januszkiewicz-Lewandowska D, Bereszyńska I, Wysocki J, et al. Interleukin 10 gene single nucleotide polymorphisms in Polish patients with chronic hepatitis C: Analysis of association with severity of disease and treatment outcome. Hum Immunol. 2017;78(2):192-200. https://doi.org/10.1016/j.humimm.2016.10.015
29.Mocellin M, de Azeredo Leitão LA, de Araújo PD, Jones MH, Stein RT, Pitrez PM, et al. Association between interleukin-10 polymorphisms and CD4+CD25+FOXP3+ T cells in asthmatic children. J Pediatr (Rio J). 2021;97(5):546-551. https://doi.org/10.1016/j.jped.2020.11.008
30.Kudo M, Melton AC, Chen C, Engler MB, Huang KE, Ren X, et al. IL-17A produced by ab T cells drives airway hyper-responsiveness in mice and enhances mouse and hu-man airway smooth muscle contraction. Nat Med. 2012;18(4):547-554. https://doi.org/10.1038/nm.2684